2017.03.15

AI創薬の革新――低コスト・低リスクで迅速な新薬開発に向けて
近年、創薬の現場では新薬開発の低迷、薬価の高騰が続き深刻な問題となっている。そんな中、注目を集めているのは人工知能(AI)の技術を利用した創薬戦略だ。AI、ビッグデータによって、医療と創薬の未来はどう変わるのか。九州大学生体防御医学研究所の山西芳裕准教授にお話を伺った。(聞き手・構成/大谷佳名)
低迷する新薬開発
――山西先生のご専門の「バイオインフォマティクス」とはどういった分野なのですか。
簡単に言えば、生命科学とITの融合分野です。近年、ゲノムを読む技術が発展し、ヒトをはじめ様々な生物種のゲノムを高速に読めるようになってきました。それに伴い、遺伝子やタンパク質の働きに関する動的なデータも計測可能になり、膨大な量のデータが生み出されるようになりました。それらをIT技術によって解析して新しい生物学的発見に繋げるのがバイオインフォマティクスという分野になります。
生命システムは、遺伝子やタンパク質、低分子化合物(分子量の小さい化合物)など、さまざまな分子が相互作用しあうことで全体としての機能を果たしています。しかし、その相互作用ネットワークの大部分はまだ分かっていません。たくさんの分子がどのように相互作用しあい、全体の生命システムを維持しているのか、また病気の発症や進行に関わっているのか。ゲノム解析や関連技術によってもたらされたビッグデータを活用し、未知の分子間相互作用ネットワークをコンピュータ上で予測することで、新しい医学的発見や医療につなげようというのが僕の研究です。
――山西先生は「AI創薬」とも言われる、人工知能の技術を利用した新薬開発にも関わられていますが、なぜ創薬においてこのような手法が求められているのでしょうか。
最近、新薬の開発が非常に難しくなってきているのです。一つの薬を作るのに約1000億円、最低でも10年以上かかると言われています。新薬開発の成功率が三万分の一というデータもあり、ほとんどが失敗に終わっているんです。失敗する理由はさまざまですが、例えば、良い薬がデザインできたとしても大量生産するのが難しい、体内での吸収や代謝が不十分、動物ではうまくいくのだけれどもヒトでは有効性が低い、最後の臨床試験で毒性が判明する、などなど。開発費用は年々増加する一方で、成功件数は減少が続いている状況なのです。
一方、今この時も病気で苦しんでいる患者さんはたくさんいるわけで、迅速な新薬開発は常に求められています。日本の場合は、特に医療費が増え続けており、さらに今後、少子高齢化社会のために医療費が増え続けるだろうという社会的な問題もあります。さらに最近は薬価も高騰しており、よく新聞やテレビでも話題になっていますが、C型肝炎の薬である「ハーボニー配合錠」は、1錠が約8万円もします。必要な新薬は作らなければならない、しかし、医療費は抑制しなければならない、非常に難しい状況にあるのです。
そこで、僕が取り組んでいるのは「ドラッグ・リポジショニング」と呼ばれる創薬戦略です。これは何かというと、すでに承認されている薬や過去に医薬品開発に失敗した化合物の新しい効能を発見し、それを本来とは別の病気の薬として開発しようというアプローチになります。そのために、近年大量に生み出されてきた医薬ビッグデータを活用し、AIの基盤技術である機械学習を駆使した手法を研究開発しています。

低コスト・低リスクで効率的な創薬
――すでに承認されている薬にも、まだ発見されていない治療効果を持っている場合があるんですね。
そうなんです。例えば有名な例ですと「シルデナフィル」、商品名の「バイアグラ
ドラッグ・リポジショニングで扱う承認薬の場合、人間の体で安全性や体内動態が確認されているので、開発途中で失敗するリスクが低いだけでなく、そのまま臨床試験でヒトに対する効果を確かめることができます。従来の医薬品開発ではゼロから作るので、長い複雑な行程があるのに対して、中間の過程をスキップできるわけです。だから、高速に、低コスト・低リスクで開発できるのが特徴です。
――既存薬の適用範囲を広げていくことで、効率の良い新薬開発が可能になるんですね。
はい。そもそも薬の効果というのは、人の体内に入って標的としているタンパク質にくっつき、そのタンパク質の機能を阻害したり、活性化させることによって生じます。しかし、本来目標としている標的タンパク質以外のタンパク質にくっついて、予期しない作用を引き起こしてしまうこともある。これが副作用が起きるメカニズムの一つです。ただ、ある薬の副作用は別の病気の患者さんにとっては効能になる場合もあるんですね。
現在、認可されている薬が数千種類、ヒトのタンパク質は数万種類、何らかの病名が付いている病気は数千種類あります。この中で、どの薬がどのタンパク質に作用し、どんな病気に効くのか、そうした未知の関係性は我々が知らないだけでたくさん眠っているはずです。意外かもしれませんが、すでに病院などで使われている承認薬でさえも、なぜ効くのか分からないものがたくさんあるんです。承認薬のうち半分以上が薬効に関与する標的タンパク質が分かっていません。ですので、承認薬を徹底的に解析することで未知の効能が見つかる可能性は高いと思います。
以前まで創薬関連のデータは非公開のものが多かったのですが、最近になって、公共のデータベースで次々に公開されるようになってきました。新薬開発を進めるために、重要なデータはシェアして有効に使っていくべきだ、という考え方が広まってきたからです。機械学習で解析できる医薬データが揃ってきたことで、ドラッグ・リポジショニングの研究は大きく躍進しています。
糖尿病の薬がパーキンソン病の治療薬にもなる?
――具体的には、どのように予測していくのですか?
我々の研究では、まず薬が作用するタンパク質を全て同定するため
もう一つは、臨床報告書の薬理作用情報を用いた手法です。患者さんが薬を飲んだ後の体の症状の変化を手掛かりに使おうというのがここでのアイデアになります。薬効や副作用などのパターンが似ている薬は同様のタンパク質に相互作用をするだろうと考えて予測をしていきます。
そして三つ目は、遺伝子発現情報を用いた手法です。薬を細胞にかけたときの遺伝子発現の変化をもとに、相互作用するタンパク質を予測する方法になります。遺伝子発現という言葉は聞き慣れないかもしれませんが、これは遺伝子がどれだけ働いているかを表します。薬に対する遺伝子の反応パターンが似ている薬は同じようなタンパク質に相互作用するだろうと考えて予測を行います。
この三つの方針のもと、それぞれ機械学習のモデルを作ります。数百万の化合物・タンパク質間相互作用のデータや様々な病気の分子機序のデータを使って予測モデルを学習させ、承認薬に適用する流れになります。
――多角的な視点から予測することで、より精度の高い結果が導き出されるんですね。
はい。大まかな手順としては、一つの薬に着目して、その薬が作用するタンパク質が分かっていなければそれを予測する、分かっている場合はそれ以外に相互作用するかもしれない他のタンパク質を予測する、この操作を繰り返していきます。様々な疾患の分子機序情報を基に、タンパク質を介してその薬を疾患に結びつけ、未知の関係性を発見していく。僕らの最近の研究では、日本や欧米における約8000個の薬に対してドラッグリポジショニングを行い、がんや神経変性疾患、免疫疾患など、約千種類の疾患に対する効能を予測しました。
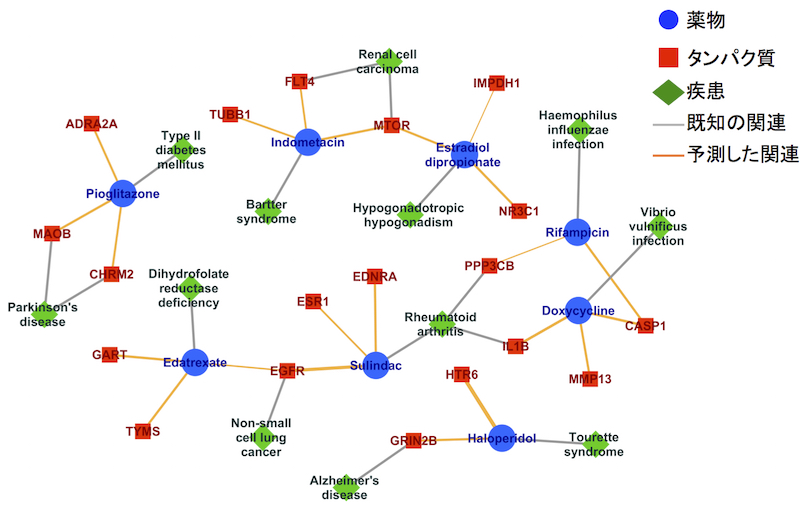
薬物・タンパク質・疾患ネットワークの一部
出典:Sawada R, Iwata H, Mizutani S, Yamanishi Y.
Target-based drug repositioning using large-scale chemical-protein interactome data. J Chem Inf Model. 55(12):2717-30, 2015.
――例えば、どのような薬の効果が予測されましたか?
化学構造情報によって予測した例として、2型糖尿病の治療薬でピオグリタゾンという薬があるのですが、これがMAOBというタンパク質と相互作用することが予測されたので、パーキンソン病にも効くのでないかと考えられます。というのも、パーキンソン病はドーパミンという神経伝達物質が減っていることが病気の原因であり、MAOBにはドーパミンを分解する酵素なので、薬によってその働きを抑えてやることで、ドーパミンの分解を防いで治療に繋げることができると考えられます。最近の論文でもこれを裏付ける報告が出てきています。実際に機械学習の学習データに入っている化合物の中で、MAOBに相互作用する化合物とピオグリタゾンは化学構造が似ていました。
遺伝子発現情報を用いた予測例としては、フェノチアジンという精神病の薬が、男性ホルモンの受容体であるARというタンパク質との相互作用が予測されたので、前立腺がんに効くのではないかと考えられる例があります。実際に実験で、ARに対する阻害作用を確認できました。そこで、どのような遺伝子の発現が応答していたかを調べると、細胞の自殺を意味するアポトーシスと呼ばれる現象に関わる遺伝子群が働いていることが分かりました。ですので、がん細胞が自滅するように誘導することで前立腺がんに効く可能性があると考えることができます。実際に、フェノチアジンの遺伝子発現パターンは、ある前立腺がんの薬と似ていました。
オーダーメイドの医療に向けて
――予測できた薬は、将来的には実際に新薬として利用できるようになるのですか。
上の例で示したものではありませんが、現在は、予測したものが実際に効果があるかを実験系研究者や臨床医学系研究者と共同研究しながら確認している段階です。探索的臨床試験までいっているものもあります。将来の製品化に向けて、複数の製薬会社と共同研究を進めています。
患者さんの手元に届けるためには製品化する必要がありますが、そのあたりのノウハウは大学の研究者は持っていないんです。さらに、保険適用になるためには治験を行わなければならないので、今後も製薬企業と連携して開発を進めていくことになると思います。
――AI技術を活用したドラッグ・リポジショニングが実用化されるようになれば、創薬はどのように変わるでしょうか。
これまでの創薬は、何万もの化合物を合成し、何百匹ものマウスや実験動物を犠牲にして成り立ってきました。こうした従来の大量消費型の創薬から、安くて効率的で、かつ省エネルギーの創薬に転換できるんじゃないかと考えています。
また、製薬会社にとってもメリットはあります。過去に作った薬や社内で眠っている化合物が他の病気にも使えることが分かれば、さらに売上を延ばすことができるからです。さらに国全体としては、新薬の価格が高騰する中、昔からある薬の場合は薬価も人件費も抑えられるので、医療費の削減にも貢献できると考えられます。
そして何より、今病気に苦しんでいる患者さんに少しでも早く薬を届けることができればと思っています。以前、世界中でエボラ出血熱が大流行しましたよね。当時も治療薬が存在せず、多くの人々が命を落としました。そんな中、ニュースでも大きく取り上げられたように、日本の富士フィルムが作っていたインフルエンザの治療薬、アビガンという薬に有効性があったことは記憶に新しいと思います。そのように、まだ治療法がない病気に対しても、迅速に必要な薬を開発できるようになることが目標ですね。
――今後の展望として、AI創薬においてはどのようなことに取り組んでいきたいですか。
そうですね。まずはより多くの病気に対して、有効性がある薬を見つけていきたいと思います。特に、まだ有効な治療法がない病気や希少疾患に対して取り組んで行きたいと思います。
あとは、個人差を考慮した創薬にも取り組んで行きたいと思います。同じ名前の病気でも人によって薬の効き具合が変わったり、病気の発症メカニズムや進行の度合いが変わったりしてきます。患者さんの表面的な情報だけでなく、ゲノム情報や他の分子情報を用いた分類を行った上で、「こういう遺伝子変異があるからこの薬を使いましょう」「こういうタンパク質の異常があるからこの薬を使いましょう」とか、個別に対応できるようになればいいなと思います。ただ個人差情報の公開データは多くないのが難しいところです。
一部の病気に関しては、現在でも薬の選択ができるようになっています。たとえば乳がんの場合、こういうタイプの人にはこの薬が効くということがだいぶ分かっているので、事前に患者さんの遺伝子情報を調べて薬を処方するというのはよく行われています。もしくは、どの遺伝子に変異が入っているのか、どの遺伝子の発現が異常かを確認して治療方針を決めるということも始められつつあります。
――それぞれ患者の特性に合った、より効果が期待できる医療につながっていきそうですね。山西さん、ありがとうございました。
プロフィール

山西芳裕
九州大学生体防御医学研究所准教授、九州大学高等研究院准教授、科学技術振興機構さきがけ研究者。京都大学大学院 理学研究科博士課程修了。パリ国立高等鉱業学校バイオインフォマティクスセンター博士研究員、京都大学化学研究所助手・助教、キュリー研究所 バイオインフォマティクスユニット常勤研究員、パリ国立高等鉱業学校バイオインフォマティクスセンター常勤研究員を経て現職。著書:Lodhi, H. and Yamanishi, Y., Chemoinformatics and Advanced Machine Learning Perspectives, IGI Global, 2010.


